English Version
English Version
|
|
|
|
PROTEÍNAS DE MEMBRANA
Las hay de dos tipos: (1) Proteínas integrales: son aquellas que cruzan la membrana y aparecen a ambos lados de la capa de fosfolípidos. La mayor parte de estas proteínas son glicoproteinas, proteínas que tienen unidos uno varios monosacáridos. La parte de carbohidrato de la molécula está siempre de cada al exterior de la célula, y (2) Proteínas periféricas: estás no se extienden a lo ancho de la bicapa sino que están unidas a las superficies interna o externa de la misma y se separan fácilmente de la misma. La naturaleza de las proteínas de membrana determina su función: Canales: proteínas integrales (generalmente glicoproteínas) que actúan como poros por los que determinadas sustancias pueden entrar o salir de la célula, Transportadoras: son proteínas que cambian de forma para dar paso a determinados productos, Receptores: son proteínas integrales que reconocen determinadas moléculas a las que se unen o fijan. Estas proteínas pueden identificar una hormona, un neurotransmisor o un nutriente que sea importante para la función celular. La molécula que se une al receptor se llama ligando, Enzimas: pueden ser integrales o periféricas y sirven para catalizar reacciones a en la superficie de la membrana, Anclajes del citolesqueleto: son proteínas periféricas que se encuentran en la parte del citosol de la membrana y que sirven para fijar los filamentos del citoesqueleto, Marcadores de la identidad de la célula: son glicoproteínas y glicolípidos característicos de cada individuo y que permiten identificar las células provenientes de otro organismo. Por ejemplo, las células sanguíneas tienen unos marcadores ABO que hacen que en una transfusión sólo sean compatibles sangres del mismo tipo. Al estar hacia el exterior las cadenas de carbohidratos de glicoproteínas y glicolípidos forman una especie de cubierta denominada glicocalix.  +info
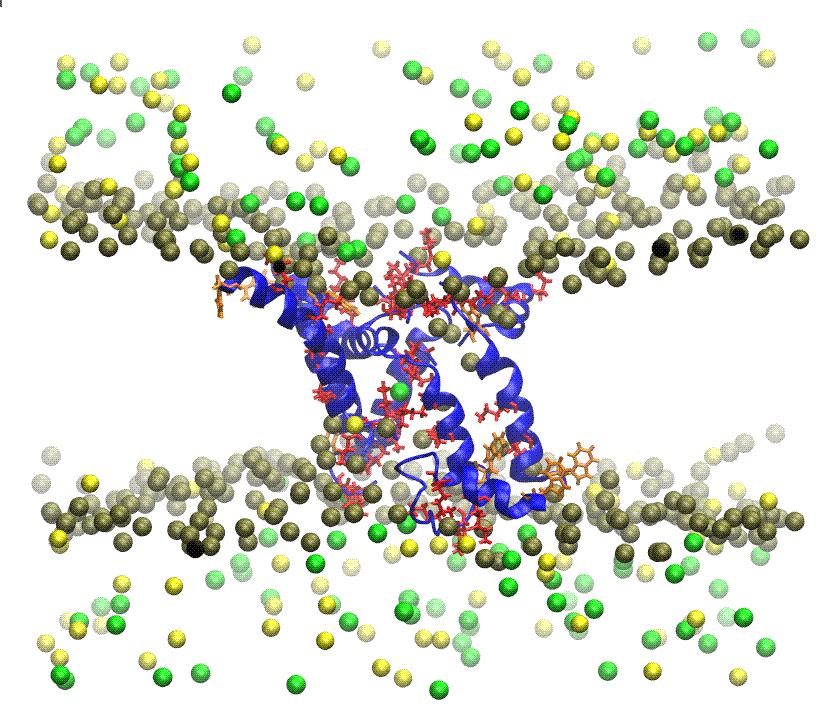
+infoPÉPTIDOS ANTIMICROBIANOS En febrero de 2017 la Organización Mundial de la Salud emitió una alerta por el uso indiscriminado de antibióticos convencionales el cual ha facilitado la resistencia de diversas bacterias a múltiples fármacos. Como la industria farmacéutica está generando cada vez menos antibióticos debido a la falta de inversión en investigaciones, y al mismo tiempo, cada vez están surgiendo más cepas de patógenos, como las superbacterias resistentes a los medicamentos actualmente disponibles, se están agotando muy de prisa las opciones terapéuticas. Los péptidos antimicrobianos permeabilizantes de membrana (PAMs) son buenos candidatos para afrontar esta crísis dado que actúan sin alta especificidad hacia un objetivo de proteína, reduciendo la probabilidad de resistencia inducida. Comprender el mecanismo de permeabilización de la membrana bacteriana es crucial para el desarrollo de PAMs como agentes antimicrobianos útiles. Con el fin de comprender el mecanismo molecular y especificidad de los PAMs con su objetivo celular, mi grupo de investigación está realizando simulaciones de dinámica molecular (DM) en modelos moleculares de complejos formados por moléculas de PAMs, aislados de los venenos de alacrán, en presencia de parches de membranas de diferente composición lipídica en disolución acuosa y en condiciones fisiológicas.  +info
+infoCANALES IÓNICOS El término canales iónicos se aplica a ciertos elementos macromoleculares, de naturaleza proteica, insertados en las membranas celulares. En las células excitables su función es producir y transducir señales eléctricas. En otro tipo de células estas proteínas transmembranales forman poros y controlan el paso de iones específicos a través de las membranas. Estos canales juegan un importante papel en la regulación del transporte transepitelial de agua y sales, y del volumen y pH celulares, y actúan como vías de señalización celular. En general los canales iónicos actúan como compuertas que se abren o se cierran en función de los estímulos externos, aunque algunas sustancias tóxicas pueden desactivar su función natural. En mi grupo de investigación estamos interesados en estudiar por medio de DM los cambios conformacionales que ocurren en los canales ionicos cuando neurotoxinas de origen animal, como las encontradas en los venenos de alacrán, ejercen su acción en canales de sodio y potasio dependientes de voltaje.  +info
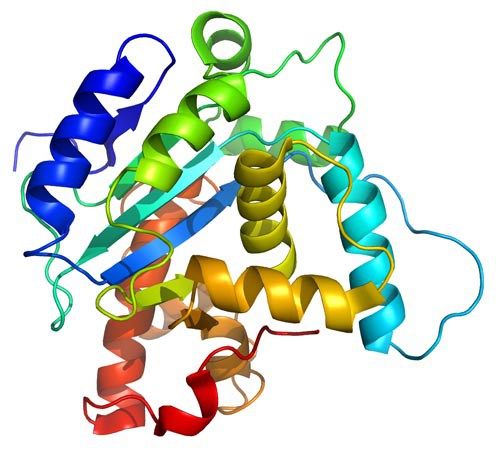
+infoPLEGADO DE PROTEÍNAS Cuando la estructura terciaria de una proteína no se ha determinado experimentalmente, se puede intentar construir un modelo tridimensional a partir de su secuencia de aminoácidos. El principio de Anfinsen afirma que toda la información requerida para especificar la estructura de una proteína está codificada en su secuencia de aminoácidos. El objetivo de la predicción de la estructura terciaria de las proteínas consiste en estimar la posición espacial de todos y cada uno de los átomos de la molécula proteica a partir de la secuencia de aminoácidos utilizando métodos computacionales. Este es uno de los retos más difíciles a los que se enfrentan los bioinformáticos y algunos lo han definido como “el santo grial de la Bioinformática”, también conocido como "El Problema del Plegado de Proteínas". Mi grupo de investigación emplea métodos ab initio, o de novo, para construir un modelo 3D de una proteína. En éstos métodos se utiliza únicamente la secuencia de amino ácidos de la proteína y basados exclusivamente en principios físicos (una función de energía) la determinación de la conformación de mínima energía, que corresponde al mínimo global de esta función, se obtiene empleando métodos de optimización numérica para problemas NP-duros tales como: Algorítmos Heurísticos. En el pasado hemos utilizado heurísticas tales somo la Búsqueda Tabu y el Recocido Simulado. Algorítmos Bioinspirados. Los algoritmos bio-inspirados son métodos de optimización basados en alguna metáfora evolutiva o por simulación de comportamientos sociales de insectos o humanos. Su uso se vuelto popular para realizar búsquedas eficientes y como técnicas de optimización debido a que son muy efectivos en la solución de problemas de optimización muy difíciles. Entre ellos tenemos a los Algoritmos Genéticos, la optimización por Colonia de Hormigas y por Enjambre de Partículas. Basados en el Conocimiento. Si bien los Potenciales de Fuerza Media en principio se pueden calcular a partir de un campo de fuerza física, la filosofía básica del enfoque Basado en el Conocimiento es aproximar estas cantidades mediante el análisis estadístico de estructuras proteicas conocidas, un enfoque a veces denominado la "ley inversa de Boltzmann". Normalmente, se usa una base de datos que contiene un conjunto de estructuras nativas conocidas a partir de datos de alta resolución, que puede incluyir una cantidad de señuelos dependiendo de la aplicación. La base de datos que se utiliza para la construcción de los potenciales basados en el conocimiento típicamente incluirá tanto variaciones estructurales como secuenciales.  +info
+infoENANTIODIFERENCIACIÓN También estamos interesados en conocer las bases moleculares del proceso de enantiodiferenciación de drogas quirales. Nuestro interés en este tema surgío cuando nos enteramos que entre 1959 y 1964 se administró la Talidomida (una molécula quiral) para evitar la ansiedad, el insomnio y las náuseas en las mujeres embarazadas. Esta droga causó terribles malformaciones de nacimiento. Solo en Europa nacieron mas de 10,000 niños gravemente deformes, muchos de ellos sin piernas ni brazos, porque sus madres habían tomado la droga al comienzo del embarazo. Posteriormente se supo que el isomero R tiene el efecto teragénico, mientras que el isomero S tiene el efecto sedativo buscado. Muchos medicamentos que hoy día se encuentran en el mercado son quirales. Para entender las bases moleculares del proceso de enantiodiferenciación nuestro grupo de investigación aplica la técnica de la DM sobre complejos de inclusión en ciclodextrinas usados como sistemas modelo de receptores quirales.  +info
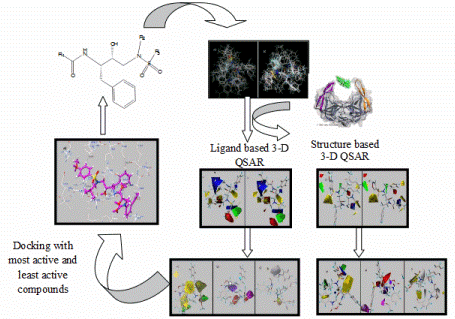
+infoQSAR Modelos cuantitativos de relación estructura-actividad (modelos QSAR) son modelos de regresión o clasificación utilizados en las ciencias químicas y biológicas y la ingeniería. Al igual que otros modelos de regresión, los modelos de regresión QSAR relacionan un conjunto de variables predictoras (X) con la potencia de la variable respuesta (Y), mientras que los modelos QSAR de clasificación relacionan las variables predictoras con un valor categórico de la variable respuesta. En el modelado QSAR, los así llamados predictores consisten en propiedades físico-químicas o descriptores moleculares teóricos de sustancias químicas. La variable de respuesta QSAR podría ser una actividad biológica de los productos químicos. Los modelos QSAR resumen primero una supuesta relación entre las estructuras químicas y la actividad biológica en un conjunto de datos de sustancias químicas. En segundo lugar, los modelos QSAR predicen las actividades de nuevos productos químicos. Mi grupo de trabajo ha incursionado en la formulación de modelos de QSAR para compuestos con diferentes propiedades biológicas como los antinociceptivos, antibacterianos, inhibidores de Sero-Albumina de Humano, inihibidores de la vasopresina y carcinógenos entre otros.  +info
+infoENERGÍA LIBRE La formación de complejos en los que participan moléculas biológicas no es un fenómeno exclusivo de la Farmacología, sino que se contempla en otras ramas de la Biología, como la Bioquímica (e.g. complejos de Michaelis entre enzimas y sustratos) o la Inmunología (e.g. complejos antígenoanticuerpo), y de la propia Química, donde las interacciones entre moléculas "huéspedes" (guests) y "hospedadoras" (hosts) han abierto nuevas perspectivas en la denominada Química Supramolecular. Cualquier acción farmacológica tiene su inicio en la formación de un complejo entre la molécula de fármaco y su sitio receptor en una macromolécula biológica. Por lo tanto, la especificidad de la respuesta a un fármaco dado viene determinada en gran medida por la capacidad de los distintos receptores celulares para reconocerlo como agonista o antagonista y evocar o no una respuesta. Para el estudio de las interacciones ligando-receptor, empleamos los métodos de la dinámica molecular para el cálculo de la energía libre de asociación (ΔG) tales como: (a) Free Energy Perturbation, (d) Umbrella Sampling, (c) Thermodynamic Perturbation, (d) Biased Sampling y sus variaciones. Para estos y otros métodos se puede consultar el libro Free Energy Computations.  +info
+info |
|
Copyright © Ramón Garduño-Juárez. All Rights Reserved. |
 E-mail: | Mayo 2017
E-mail: | Mayo 2017